本記事は、kaggle Advent Calendar 2018の11日目の記事です。
執筆のきっかけ
先日参加したKaggle Tokyo Meetup #5 の ikiri_DS の発表「Home Credit Default Risk - 2nd place solutions -」にて、遺伝的プログラミングで生成した特徴がLocal CV、Public LB、Private LBの全てで精度向上に貢献したという話がありました。
遺伝的プログラミングや遺伝的アルゴリズムは、大学の学部時代から興味があり、ブログでも何度か取り上げてきました。しかしKaggleなどで試したことはなかったので、自分で手を動かして検証してみようと考えた次第です。
遺伝的プログラミングによる特徴量生成
遺伝的プログラミングによる特徴量生成の理論的な背景や実装については、昨年に執筆された下記の記事が非常によくまとまっています。
ただし本記事では、Kaggleに用いるという観点から、この手法で生成した特徴量がKaggleでよく使われる分類器「LightGBM」でも効果を発揮するか分からない点が気になりました。
本記事では、この点について実験で検証したいと思います。
LightGBMによる実験
実装はGitHubで公開したので、興味ある方はご参照ください。
最初に、実験に用いるデータセットを作成します。特徴量が10個ある10000行のDataFrameを作成しました。20%をtestデータとして分割しておきます。
from sklearn.datasets import make_classification from sklearn.model_selection import train_test_split # サンプルデータの生成 X, y = make_classification(n_samples=10000, n_features=10, n_informative=5, n_redundant=0, n_repeated=0, n_clusters_per_class=8, random_state=123) X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=123) # LightGBM用の分割 X_train2, X_valid, y_train2, y_valid = train_test_split(X_train, y_train, test_size=0.2, random_state=123)
特徴量生成前
まずは単純にLightGBMで精度を検証してみます。
import lightgbm as lgb import matplotlib.pyplot as plt lgb_train = lgb.Dataset(X_train2, y_train2) lgb_eval = lgb.Dataset(X_valid, y_valid, reference=lgb_train) lgbm_params = { 'learning_rate': 0.1, 'num_leaves': 8, 'boosting_type' : 'gbdt', 'reg_alpha' : 1, 'reg_lambda' : 1, 'objective': 'regression', 'metric': 'auc', } # 上記のパラメータでモデルを学習する model = lgb.train(lgbm_params, lgb_train, # モデルの評価用データを渡す valid_sets=lgb_eval, # 最大で 1000 ラウンドまで学習する num_boost_round=1000, # 10 ラウンド経過しても性能が向上しないときは学習を打ち切る early_stopping_rounds=10) # 特徴量の重要度をプロットする lgb.plot_importance(model, figsize=(20, 10)) plt.show()
[1] valid_0's auc: 0.743222 Training until validation scores don't improve for 10 rounds. [2] valid_0's auc: 0.774293 [3] valid_0's auc: 0.786269 [4] valid_0's auc: 0.796968 [5] valid_0's auc: 0.798784 (中略) [172] valid_0's auc: 0.855702 [173] valid_0's auc: 0.855937 [174] valid_0's auc: 0.85584 Early stopping, best iteration is: [164] valid_0's auc: 0.856409
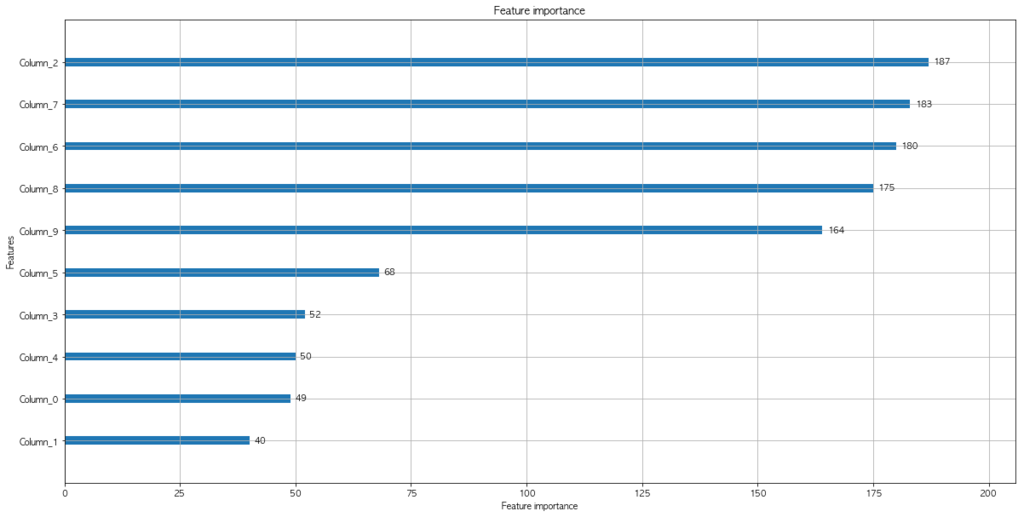
testデータで確認したところ、AUC=0.854653164841を得ました。
from sklearn.metrics import roc_auc_score # テストデータを予測する y_pred = model.predict(X_test, num_iteration=model.best_iteration) # auc を計算する auc = roc_auc_score(y_test, y_pred) print(auc)
特徴量の生成
遺伝的プログラミングを用いた特徴量の生成に関しては、先の記事からほとんど変更していません。ベースのモデルはL2正則化ロジスティック回帰、評価方法はtrainデータの5-foldクロスバリデーションAUCを利用しています。
ここでのベースのモデルもLightGBMなどの勾配ブースティング系に変更しようと試してみましたが、計算量が膨大になってしまうため、比較的単純なモデルのままとしました。
生成された特徴量は以下の通りです。
### Generated feature expression mul(sub(add(mul(ARG9, ARG2), cos(neg(ARG6))), neg(ARG6)), sub(ARG7, cos(cos(neg(ARG6))))) mul(neg(ARG10), mul(ARG9, ARG8)) sub(add(ARG1, ARG8), add(mul(add(ARG2, ARG6), ARG8), cos(protectedDiv(ARG10, ARG3)))) sub(sub(sub(neg(mul(ARG9, ARG12)), cos(ARG9)), cos(ARG9)), mul(ARG2, ARG12)) mul(add(add(ARG6, neg(ARG7)), ARG8), add(sin(ARG6), neg(ARG7))) sin(cos(add(ARG8, ARG2))) cos(cos(add(cos(ARG9), sin(sin(ARG2))))) mul(sub(ARG10, mul(mul(ARG7, ARG2), cos(ARG6))), ARG16) mul(ARG14, ARG8) mul(sub(sub(ARG14, ARG9), sin(ARG6)), add(ARG9, ARG2)) mul(ARG11, cos(ARG16)) sin(cos(ARG7)) mul(add(cos(ARG9), tan(ARG21)), ARG8) cos(ARG22) mul(sub(ARG23, ARG21), sub(sin(ARG9), add(sub(sin(ARG9), add(ARG13, ARG3)), ARG3))) mul(add(add(ARG21, neg(neg(ARG16))), ARG2), ARG23) mul(ARG2, neg(mul(ARG9, ARG9))) mul(cos(ARG14), ARG26) sub(mul(mul(cos(ARG13), ARG0), ARG26), mul(ARG17, ARG26)) mul(cos(ARG6), ARG2)
例えば「mul(cos(ARG6), ARG2)」は、「cos(特徴量6) × 特徴量2」という式で計算される特徴量を意味します。生成された特徴量を眺めてみると、人間ではなかなか思いつかないような式が生成されていると分かります。
10個の特徴量から新たに20個の特徴量を生成するのに、下記スペックのMacBook Proで1時間30分程度かかりました。元にする特徴量の数やDataFrameの行数によっては、かなりの時間がかかると見込まれます。
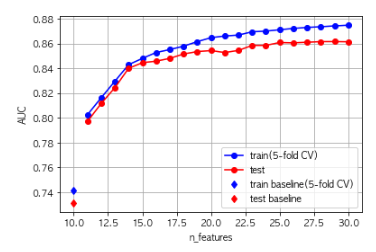
またtrainデータとtestデータに関する精度は、次の図のように向上していきました。
特徴量生成後
特徴量生成前と同様に、LightGBMで精度を検証しました。
[1] valid_0's auc: 0.752588 Training until validation scores don't improve for 10 rounds. [2] valid_0's auc: 0.770969 [3] valid_0's auc: 0.791902 [4] valid_0's auc: 0.796553 [5] valid_0's auc: 0.79827 (中略) [137] valid_0's auc: 0.866912 [138] valid_0's auc: 0.866721 [139] valid_0's auc: 0.866903 Early stopping, best iteration is: [129] valid_0's auc: 0.867409
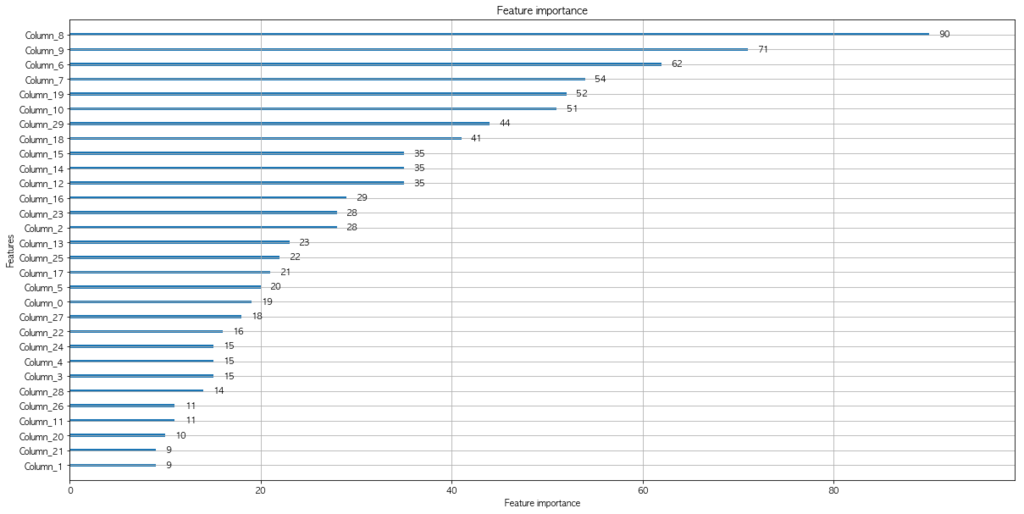
testデータで確認したところ、AUC=0.877752818887 (> 0.854653164841)と、特徴量を加える前より高い精度を示しました。
特徴量の重要度を確認したところ、上位5番目から新規に追加した特徴量(インデックスが10以上のもの)が登場しています。
おわりに
本記事では、遺伝的プログラミングによる特徴量生成について、この手法で生成した特徴量がLightGBMを用いた場合でも効果を発揮すると確認しました。
ただし最大のボトルネックとして、計算時間の問題が残ります。遺伝的プログラミングでは大量の試行錯誤を実施するため、今回のさほど大きくないデータセットでも一定の時間が必要となりました。実際のコンペに投入するに当たっては、事前に特徴選択を実施するなどの対応が必要かもしれません。